Définition Modification directe et dirigée du génome d’un organisme : bio-ingénierie. Biotechnologie: utilisation des voies biochimiques et métaboliques d’un organisme pour la production industrielle. Par exemple, les diabétiques doivent prendre de l’insuline et au début, cette insuline a été produite à partir d’insuline de porc, mais cette dernière a du par la suite être modifiée pour être mieux tolérée par les patients.
Procédés de Bio-ingénierie
Hybridation Des outils génétiques Analyse de l’ADN (électrophorèse et PCR) Technologie de l’ADN recombinant Thérapie génique Analyse du génome (empreintes digitales et puces à ADN)
OUTILS GENETIQUES
Enzymes pour la découpe, le collage, l’inversion des acides nucléiques. Permet à l’analyse de l’ADN, en utilisant les enzymes comme des outils: 1) ADN hélicase 2) Ligase : joints scellent deux morceaux d’ADN ensemble comme une colle moléculaire. 3) Polymerase : Lit un brin d’ADN et synthétise un brin complémentaire. Les ADN polymérases lient et répliquent des brins d’ADN. Les ARN polymérases peuvent à partir de brins d’ADN synthétiser des brins d’ARN. 4) La transcriptase inverse (fait des copies d’ADN à partir d’ARN) : crée une copie d’ADN à partir d’un brin d’ARN. C’est une propriété très inhabituelle et c’est la raison pour laquelle il est appelé transcriptase inverse. (puisque la transcription aboutit normalement à la formation d’ARN à partir d’ADN). Cette enzyme ne se trouve pas dans toutes les cellules; il se trouve que dans un couple de virus. Seule une petite une famille de virus VIH possède cette enzyme. 5) Les endonucléases de restriction reconnaissent des séquences d’ADN spécifiques et les découpent 6) Hélicase: déroule l’ADN double brin et vous pouvez le faire dans le tube à essai et l’autre possibilité est la chaleur qui dénature également l’ADN double brin; Imaginez une séquence d’ADN, et nous découpons cette dernière avec trois endonucléases de restriction différentes. Nous aurons ainsi des fragments. 

Si nous avons deux enzymes avec 2 sites de coupure, nous obtiendrons trois fragments. Après cela nous pouvons les mettre sur un gel d’électrophorèse, procédé qui permet de séparer des fragments d’ADN en fonction de leur taille: Comment ça marche? Nous prenons un gel et nous mettons nos échantillons en haut de gel, puis nous l’exposons à une charge (normalement ADN a une charge négative, car il a de nombreux groupes de phosphate). Ainsi l’ADN commence à se déplacer vers le charge positive. La rapidité du déplacement dépend de la taille des fragments. Les grands fragments se déplacent plus lentement que les petits fragments; de sorte qu’il sépare les fragments d’ADN en fonction de leur taille. Nous allons voir un exemple avec une séquence d’ADN. Vous prenez un morceau d’ADN et nous souhaitons vérifier la séquence pour voir si elle correspond au gène vous pensez que c’est… Où est la séquence d’ADN que vous attendez? 5 ‘AAGCTAAGGATTCGTAT 3′ 3 ‘TTCGATTCCTAAGCATA 5′ Et vous pensez savoir où est cet ADN mais vous voulez vous assurer que c’est le gène correspondant. Ce que vous pouvez faire est de vous couper cet ADN par des endonucléases de restriction et vous savez que l’enzyme 1 coupe après la séquence AGCT. Et l’ enzyme 2 coupe après la séquence AGGA. Si vous ajoutez ces deux endonucléases de restriction différentes pour cet ADN vous pouvez prévoir combien de fragments vous allez trouver: 5 ‘AAGCTAAGGATTCGTAT 3′ 3 ‘TTCGATTCCTAAGCATA 5′ Nous devons trouver trois fragments après la procédure (après digestion complète), mais vous pouvez également connaître la taille de chacun de ces fragments. Vous savez que ce premier fragment doit être seulement trois nucléotides de long et le second fragment devrait être cinq nucléotides de long et le troisième fragment doit être neuf nucléotides de long. Maintenant, comment nous pouvons voir la taille des fragments? Vous allez l’exécuter sur gel d’électrophorèse. Maintenant, vous mettez votre échantillon sur la surface du gel (après digestion), vous devez ajouter un mélange standard de chaque côte. Ensuite, vous exposez 1+1 à une charge. En combien de temps se déplace chaque fragment d’ADN dépend de sa taille, des fragments plus petits se déplacent plus vite, les fragments plus gros se déplacent plus lentement. Ainsi vous obtenez le mélange standard où vous avez un nucléotide, 2 fragments de nucléotides. Dans notre expérience, nous nous attendons à 3 fragments en position 3-5-9. 
Donc, des endonucléases de restriction peuvent être utilisés comme un moyen de vérification d’une séquence. Si vous avez un morceau d’ADN et vous n’êtes pas à 100 % sûr que vous voulez juste faire une double vérification avant de faire une autre manipulation.
Réaction en chaîne par polymérase (PCR)
PCR amplifie l’ADN: il est comme une machine de copie de l’ADN PCR polymérise des copies d’ADN par une réaction en chaîne, ce qui est important parce que souvent vous pouvez avoir un échantillon d’ADN, mais vous n’en avez pas assez pour pouvoir l’analyser.
Juste parler, nous allons faire une électrophorèse sur gel ……… Nous aurons besoin d’une quantité considérable d’ADN pour exécuter ces différents tests. Très souvent, le problème est simplement que nous n’avons pas suffisamment d’ADN. Ainsi, la réaction en chaîne de la polymérase qui est une machine de copie peut nous aider à produire assez d’ADN dont nous avons besoin. Nous pouvons obtenir des millions de fragments en 4-6 heures Comment ça marche? Ici, nous avons un tube de PCR. Maintenant, si nous voulons une copie d’un morceau d’ADN Voyons voir ce genre de choses, nous devons lancer le tube de réaction. Les composants nécessaires sont : -une séquence d’ADN – un ADN-polymérase – des amorces – mélange nucléotidique Et puis nous ajoutons notre tube de PCR à une machine PCR
Et la machine de PCR modifie la température afin de pouvoir copier l’ADN. Ici, nous avons notre modèle, une machine PCR qui monte jusqu’à 94 ° (ce qui est assez élevé pour dénaturer l’ADN et les deux brins se séparent). Puis l’appareil passe à 55 ° (température à laquelle les amorces peuvent se fixer), la machine passe ensuite à 72 ° (à cette température l’ ADN polymérase attache les amorces et commence à les copier pour obtenir en bout du compte deux exemplaires. Puis la machine remonte à 94 ° (…. « » « » « » 55 ° (….. « » « » « » 72 ° (…. Et ……..le processus se répète encore et encore de façon à obtenir une quantité d’ADN supérieure grâce à……. température.
Avant l’apparition du de PCR on passait par les incubateurs (un à 94 °, un à 55 ° et un 72 °).
Récapitulatif des méthodes d’analyse de l’ADN
1) Les enzymes peuvent être utilisés comme des outils moléculaires 2) Le gel d’électrophorèse: sépare les fragments d’ADN selon leur taille 3) la réaction de polymérisation en chaîne: – Amplificateurs ADN (faire des copies de l’ADN) – Une bonne conception d’amorces permet l’isolement et l’amplification d’un gène particulier sur l’ensemble du génome En utilisant le PCR pour isoler et amplifier un gène du génome – Les amorces sont conçues pour se lier un lieu précis en fonction de règles de complémentarité des bases d’ADN – Comme l’ADN polymérase III ne peut démarrer la réplication de l’ADN où l’amorce est fixée le site de fixation de l’amorce contrôle que l’ADN est répliqué par le PCR. – En concevant des amorces complémentaires sur chaque côté du gène d’intérêt, vous pouvez spécifier la PCR pour isoler et amplifier juste ce gène et rien d’autre 1) Isoler et amplifier une séquence spécifique à partir d’un morceau d’ADN 2) ici est un morceau d’ADN du génome humain
Le clonage
I Définition
Clonage : Transfert transitoire ou stable d’un ADN étranger dans une cellule procaryote ou eucaryote
Utilisation de vecteurs : plasmides, bactériophages, virus, cosmides …
II Applications
1)Biotechnologies
Pour la production de protéines eucaryotes par des micro-organismes recombinants tels que les hormones (insuline), vaccins ou anticorps.
2)Thérapie génique
Transfert d’un gène chez l’Homme pour prévenir l’apparition ou ralentir une maladie
3) Médecine légale
Empreinte digitale empreinte génomique
Clonage plasmidique
1) Les plasmides
Ce sont des petites molécules d’ADN bicaténaires, circulaires, extra chromosomiques qui ont la capacité de se répliquer de façon autonome.
Elles sont présentes naturellement dans le cytoplasme de nombreuses souches bactériennes. Ce qui génère d’ailleurs la résistance bactérienne aux antibiotiques.
Ces plasmides sont utilisés en génie génétique pour :
– le clonage et l’amplification d’une séquence d’ADN
– l’introduction d’un gène dans des bactéries (transformation), des cellules animales (transfection) ou des organismes entiers (animaux transgéniques).
– la production de protéines à l’échelle industrielle
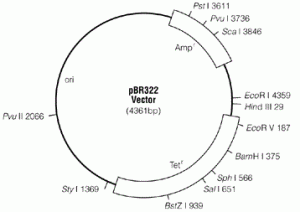
Plasmide pBR322
pBR322 contient 4361 paires de bases et contient le réplicon du plasmide pMB1, le gène ampR, codant pour la protéine de résistance à l’ampicilline (source plasmide RSF2124) et le gène tetR, codant pour la protéine de résistance à la tetracycline. Le plasmide possède des sites de restriction uniques pour plus de quarante enzymes de restriction. 11 de ces 40 sites se trouvent dans le gène tetR. Il existe deux sites pour les enzymes de restriction HindIII et ClaI au sein du promoteur du gène tetR. Il ya six sites de restriction clés à l’intérieur du gène ampR. L’origine de réplication est un site ori.
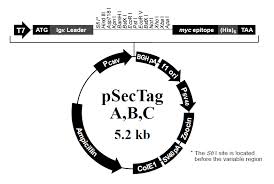
Plasmide pSecTag
L’insert
– T7 : fixe l’ARN polymérase
– ATG : codon initiateur (transcription)
– Igkappa leader : permet la sécrétion
– myc : détection de la protéine
– (His)6 : purification et détection
– TAA : terminaison de la transcription
Le plasmide
– PCMV : expression de la protéine
– BGH : facilite le séquençage
– f1 ori : réplication de l’ADN (bactéries)
– PSV40 : expression de la résistance zéocine
– Hygromycine : sélection (cellules mammifères)
– SV40 pA : terminaison de la transcription
– pUC : favorise réplication (bactéries)
– Ampicilline : sélection (bactéries)
Les enzymes de restrictions
Pour 1 digestion enzymatique : – H20 qsp 20 μl
– Tampon 10X (spécifique de chaque enzyme) : 2 μl
– BSA 0,2 μl
– ADN : 1 μl
– Enzyme : 1,5 μl —> 1h à 37°C
Migration sur gel d’agarose 0,5%