Biologie 1ere annee universitaire base
- Définition
La réplication de l’ADN est l’un des principaux mécanismes du cycle cellulaire. C’ est un processus biologique très régulé au cours duquel sont mises en place des mécanismes de réparation pour assurer l’intégrité du génome.
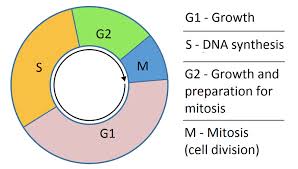
La phase S ou phase de réplication suit la phase G1 pendant laquelle la cellule a synthétisé tous les éléments nécessaires à cette réplication. La duplication de l’ADN dure environ 8 heures.
- Acteurs de la réplication
2.1. Brin matrice
La double hélice d’ADN est séparée en 2 brins d’ADN parents. Chaque brin sert alors de matrice pour la synthèse d’un nouveau brin ou brin fils.
La réplication produit ainsi 2 molécules hybrides formées d’un brin parent et d’un brin fils: la réplication est semi conservatrice.
2.2. Nucléotides
- Nucléotides: dATP, dCTP, dTTP apportant à la fois :
- le substrat : le nucléoside monophosphate,
- l’énergie pour relier les nucléotides entre eux.
2.3. Enzymes
- Il existe plusieurs ADN polymérases selon leur activité réplicative :
- alpha : début de la réplication
- delta : réplication du génome nucléaire
- gamma : réplication du génome mitochondrial
- beta : réparation de l’ADN
- epsilon : réplication des télomères
- D’autres protéines sont également nécessaires lors de la réplication
- Mécanisme de la réplication
3.1. Origines de réplication
Chez les eucaryotes, la réplication est initiée à plusieurs origines de réplication appelées OriC, contrairement aux procaryotes chez qui il n’existe qu’une seule origine de replication.
Les cellules eucaryotes doivent répliquer à chaque cycle cellulaire la totalité de leur génome. Afin de réaliser ce processus en un temps record, l’initiation de la réplication au cours de la phase S du cycle cellulaire se fait au niveau de plusieurs OriC ou « origines de réplication » le long du chromosome (environ 10000) dans des unités de réplications indépendantes appelées réplicons (30 à 300 pb).
Chez les Eucaryotes, les origines de réplication les mieux caractérisées sont celles de la levure. Elles sont nommées ARS pour Autonomously Replicating Sequence. Ces régions ARS, riches en AT, possèdent des sites de fixation pour les complexes ORC.
Ensuite seront recrutées d’autres protéines capables de dérouler la double hélice d’ADN (par activation des hélicases).
3.2. Activation des origines de réplication
3.2.1. Formation du complexe de pré-réplication
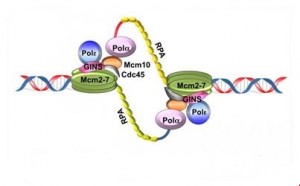
Les origines de réplication sont reconnues par des complexes protéiques « ORC » (Complexe de Reconnaissance de l’Origine) formées de six sous-unités dont chacune se fixe sur chaque origine de réplication.
Suite à cette fixation, deux autres facteurs protéiques, Cdc6/Cdc18 et Cdt1, rejoignent le premier complexe pour le recrutement de six complexes protéiques Mcm2-7 (Mcm2, 3, 5, 6, 7, 8).
3.2.2. Activation du complexe pré-RC
- Activation des CDK
- phosphorylation/ activation des protéines dont les hélicases Mcm2-7
- hélicases déroulent la double hélice d’ADN et séparent les 2 brins
Le déroulement de l’ADN et la séparation des 2 brins font apparaître des régions d’ADN simple brin, stabilisées par des protéines RPA (protéines de réplications A), accessibles aux enzymes et protéines nécessaires à la réplication.
4.recrutement des ADN polymérases et autres protéines
3.3. Les ADN polymérases
Les ADN polymérases (ou désoxynucléotidyl-transférase) sont les enzymes responsables de la polymérisation des nucléotides lors de la réplication de l’ADN. Les ADN polymérases procaryotes sont de 3 types (I, II et III) et les ADN polymérases eucaryotes de 5 types (α, β, δ, ε et γ).
3.3.1 L’ADN polymérase α
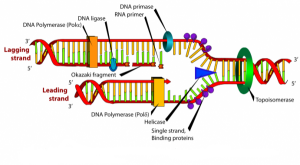
L’ADNpol α – primase synthétise l’amorce (= primer) qui est un court segment d’ARN (10 paires de bases). Ces courts fragments d’ARN sont ensuite allongés par un fragment d’ADN adjacent de 20 à 40 nucléotides de long. Une amorce ARN-ADN est ainsi créée.
3.3.2. L’ADN polymérase δ
La synthèse de l’ADN se fait toujours dans le sens 5’ vers 3’, ceci nécessite donc la présence d’un brin précoce (ou primaire) qui est le brin lu dans le sens de la fourche et d’un brin tardif (ou secondaire) qui est le brin lu dans le sens inverse de la fourche et qui est dit brin discontinu. On parle ainsi de réplication semi-discontinue.
Un complexe RF-C/PCNA (facteur de réplication C-antigène nucléaire de prolifération cellulaire) se fixe sur l’extrémité 3’OH de cette amorce ARN/ADN néosynthétisée, dissocie l’ADN polymérase α de la matrice d’ADN, laissant la place à l’ADN polymérase δ qui reconnaît le complexe RFC/ PCNA et sera responsable de la synthèse du brin continu.
Sur le brin à synthèse discontinue, l’ADN polymérase α-primase préalablement dissociée remet en route la synthèse d’une nouvelle amorce d’ARN/ADN. Sur ce brin d’ADN 5′ → 3′, l’enzyme synthétise de multiples petits fragments d’ADN à la suite des amorce, ce sont les « fragments d’Okazaki » (long d’environ 1000 à 5000 nucléotides).
Au fur et à mesure de la progression de la fourche de réplication :
- des hélicases déroulent la double hélice d’ADN,
- des topo- isomérases diminuent les torsions de l’ADN en amont de ces fourches.
3.3.3. L’ADN ligase
Elle reconstitue la liaison phosphoester entre le carbone 3′-OH et le phosphate-5′ de deux nucléotides voisins sur un brin de DNA.
Elle intervient aussi dans de nombreux processus de réparation de l’ADN.
3.4 Les enzymes topo-isomérases
- Type I
– réalise une coupure sur l’un des brins de l’ADN.
– transitoire
– relâche la torsion.
– pas d’ATP nécessaire.
- Type I I
– réalise une coupure sur les deux brins de l’ADN.
– ATP nécessaire.